Books
Projects
Citation
Editorships
Aprendizado de Máquina
| Aulas | Links |
|---|---|
|
Apresentação da Disciplina Aprendizado de Máquina
Autor: Dr. Walter F. de Azevedo Jr.
Aprendizado de máquina representa uma das áreas mais ativas de pesquisa e desenvolvimento desta geração. Seu amplo leque de aplicações e seu rápido desenvolvimento atrai diversos interessados, desenvolvedores e usuários de suas técnicas. Os métodos de aprendizado de máquina aliados ao crescimento exponencial da geração de dados científicos possibilitam aos cientistas de todas as áreas criar modelos computacionais, bem como analisar quantidades de dados que eram impensáveis somente uma década atrás. A presente disciplina tem como objetivo aumentar o número de cientistas que usam aprendizado de máquina na sua pesquisa. Apresentarei durante o semestre as principais técnicas, destacando sempre as aplicações científicas. Não há requisitos para esta disciplina, além da curiosidade científica e a mente aberta para o novo. Boas-vindas para o novo... Que a luz da ciência acabe com as trevas do negacionismo. Walter F. de Azevedo, Jr. Palavras-chave: Aprendizado de máquina; aprendizado de máquina supervisionado; conjunto de treinamento; conjunto de teste; instância; feature; variável rótulo; variável alvo; modelagem de sistemas; sistemas complexos; complexidade; sistemas biológicos; pesquisa científica; pesquisa interdisciplinar; interdisciplinaridade. |

|
|
O Método Científico
Autor: Dr. Walter F. de Azevedo Jr.
Aqui apresento os principais conceitos do método científico. Na minha visão, o ensino do método científico é negligenciado e desvalorizado no ensino superior no Brasil. Muitos professores delegam o seu ensino às disciplinas de humanas, onde boa parte dos que ensinam estão distantes da prática científica. Não abro mão de ensinar a todos meus estudantes os fundamentos do método científico. Ignorar o método científico gera negacionismo. Ou pior, a indiferença quanto ao negacionismo gera passividade, onde abrimos a porta para que os negacionistas tomem o poder. O negacionismo no poder leva ao terraplanismo, à negação da eficácia das vacinas e à desvalorização das conquistas da ciência e tecnologia. Vimos e vemos de forma constante o ataque à eficácia das vacinas e a relativização da verdade, onde dizem que a verdade científica é só mais uma das opiniões. A evolução não é uma opinião é um fato. O céu é azul não por vontade de uma criatura mística e sim pelo espalhamento Rayleigh. Ler horóscopo é compactuar com a pseudociência. Não é uma atitude ingênua e inofensiva. Um clique dado a um site de horóscopo gera lucro para grupos que enriquecem enganando. Horóscopo é mais uma porta de entrada dos negacionistas para se manterem ativos e remunerados propagando a pseudociência. Aqui também descrevo a composição de um artigo científico e apresento alguns índices de métricas científicas como o índice h e o quociente m. Discuto como a comunidade científica avalia o impacto da pesquisa por meio das métricas apresentadas. Apresento o trabalho de Ioannidis et al. (Ioannidis JPA, Boyack KW, Baas J. Updated science-wide author databases of standardized citation indicators. PLoS Biol. 2020 Oct 16;18(10):e3000918. doi: 10.1371/journal.pbio.3000918. PMID: 33064726; PMCID: PMC7567353) que estabelece métricas para avaliar o impacto da pesquisa científica em todas as áreas do conhecimento humano. Boa parte da pesquisa científica é mantida com recursos públicos. O uso eficiente dos recursos públicos deve levar a uma pesquisa científica de impacto e que gere retorno para a sociedade que a sustenta. Pesquisa científica sem impacto científico não é pesquisa é perpetuação da mediocridade. Palavras-chave: Física; física matemática; pesquisa científica; pesquisa interdisciplinar; interdisciplinaridade; educar pela ciência |

|
|
Aula Magna: Aprendizado de Máquina para o Estudo de Interações Intermoleculares.
Autor: Dr. Walter F. de Azevedo Jr.
Nesta aula magna apresento parte das minhas pesquisas focadas na modelagem das interações intermoleculares de fármacos com proteínas. Uso abordagens de aprendizado de máquina para gerar modelos computacionais que empregam as coordenadas atômicas de complexos proteína-ligante para calcular a afinidade deste ligante pela proteína. Palavras-chave: Aprendizado de máquina; aprendizado de máquina supervisionado; conjunto de treinamento; conjunto de teste; instância; feature; variável rótulo; variável alvo; modelagem de sistemas; sistemas complexos; complexidade; sistemas biológicos; pesquisa científica; pesquisa interdisciplinar; interdisciplinaridade. |

|
|
Síncrotron
Autor: Dr. Walter F. de Azevedo Jr.
Radiações têm papel bem conhecido para geração de imagens médicas, sendo a radiação X a mais utilizada. Nesta videaula de hoje veremos como podemos produzir raios X para diagnóstico e pesquisa básica sobre macromoléculas biológicas. Descreveremos a forma convencional de produção de raios X por meio de um tubo de raios X e através de um equipamento chamado sincrotron. Este usa um acelerador de partículas para a geração de radiação. Destacaremos as aplicações no estudo de possíveis fármacos. Palavras-chave: Física Computacional; física; biofísica, moléculas; biologia; biotecnologia; nanotecnologia; proteínas; raios X; produção de raios X; cristalografia; síncrotron; radiação síncrotron; Laboratório Nacional de Luz síncrotron; Laboratório Sirius; desenvolvimento de fármacos. |

|
|
Modelagem Molecular
Autor: Dr. Walter F. de Azevedo Jr.
Podemos usar abordagens da física básica para criarmos modelos computacionais para o estudo de moléculas, entre elas moléculas de proteínas. É possível considerar as interações entre os átomos de uma proteína como um sistema massa-mola, onde consideramos a ligação covalente entre dois átomos como se fosse mediada por uma mola. Quando não temos a estrutura tridimensional de uma proteína determinada experimentalmente, podemos gerar modelos a partir de uma abordagem computacional chamada modelagem molecular por homologia. Destacaremos as aplicações dessa abordagem computacional e como podemos integrá-la no desenvolvimento de fármacos. Palavras-chave: Biofísica; física; física computacional; biologia de sistemas; bioinformática; aminoácidos; proteínas; energia potencial; descoberta de fármacos; modelagem molecular; docagem molecular. |
|
|
Inteligência Artificial
Autor: Dr. Walter F. de Azevedo Jr.
Inteligência artificial tem grande potencial de gerar modelos computacionais para o estudo da vida. Especificamente, uma subárea da inteligência artificial chamada de aprendizado de máquina tem se mostrado útil na modelagem de sistemas biológicos. Dados genômicos, proteômicos, metabolômicos entre outros fornecem uma base sólida que pode ser usada para gerar previsões do comportamento de sistemas biológicos. Especificamente para descoberta de fármacos, informação sobre estrutura tridimensional junto com dados de afinidade de ligantes por proteínas nos permitem gerar modelos de aprendizado de máquina que estimam interação proteína-ligante. Veremos como usar essas abordagens no estudo de novos fármacos. Uma grande parte dos cientistas influentes mundialmente acredita que vivemos um momento especial da história do desenvolvimento científico e tecnológico. Devido à importância deste momento, destaco nos meus cursos e palestras alguns aspectos relevantes da singularidade tecnológica. Hoje, discutiremos o aumento da expectativa de vida e a possibilidade de atingirmos a singularidade tecnológica. Considerando-se o crescimento constante do poder de processamento dos computadores, espera-se que num futuro próximo sejamos capazes de transferir nossa consciência para computadores com complexidade do cérebro humano. Ficção científica ou realidade? Palavras-chave: Inteligência artificial; aprendizado de máquina; machine learning; deep learning; modelo de aprendizado de máquina; biologia de sistemas; bioinformática; biologia de sistemas; singularidade tecnológica; descoberta de fármacos; interação proteína-ligante; moléculas; aumento da expectativa de vida; transplante de órgãos; xenotransplante; transhumanismo; lei de Moore; singularidade tecnológica. |

|
|
Projetos de Pesquisa em Aprendizado de Máquina
Autor: Dr. Walter F. de Azevedo Jr.
Nesta aula temos uma descrição de três proteínas que podem ser estudadas a partir de abordagens de aprendizado de máquina. Para todas as três proteínas, temos dados sobre a estrutura tridimensional e informações sobre a interação de ligantes. A fusão desses dois dados permite que elaboremos modelos de aprendizado de máquina que façam previsões se uma dada molécula tem potencial de ser um fármaco. A proteínas são as seguintes: quinase dependente de ciclina, purina nucleosídeo fosforilase e acetilcolina esterase. Palavras-chave: Aprendizado de máquina; modelo de aprendizado de máquina; interação proteína-ligante; docking; molecular docking; docagem; docagem molecular; p53; p21; cyclin-dependent kinase 2; CDK2; roscovitina; câncer; fármacos; inibidores; bolsão de ligação de ATP; Molegro Virtual Docker; purina nucleosídeo fosforilase; modulação da resposta imunológica; acetilcolina esterase; mal de Alzheimer; cloridrato de donepezila. |

|
|
Aprendizado de Máquina Supervisionado I
Autor: Dr. Walter F. de Azevedo Jr.
Nesta aula veremos como usar as características (features) da interação entre ligantes e uma proteína alvo para gerarmos um modelo de aprendizado de máquina supervisionado que prevê se o ligante inibe a proteína. Usando a analogia da chave e fechadura, o nosso modelo será treinado com diferentes chaves que se ligam na mesma fechadura. O modelo de aprendizado de máquina supervisionado irá prever se uma nova chave se encaixa ou não na fechadura. Na analogia o “encaixar da chave” representa a inibição da proteína. Essa abordagem tem grande potencial na descoberta de fármacos, pois uma vez treinado um modelo de aprendizado de máquina, podemos testar milhões de moléculas e focar os testes pré-clínicos e clínicos nas moléculas que tiveram melhores resultados previstos pelo modelo de aprendizado de máquina. O foco da aula é no estudo de inibidores da proteína quinase dependente de ciclina 2 (CDK2), onde iremos gerar modelos de regressão linear múltipla. Palavras-chave: aprendizado de máquina; machine learning; modelo de aprendizado de máquina; biologia de sistemas; bioinformática; descoberta de fármacos; interação proteína-ligante; modelo chave-fechadura; quinase dependente de ciclina; CDK2; Molegro Data Modeller; regressão linear; regressão linear múltipla; predição; concentração inibitória a 50 %; IC50. |

|
|
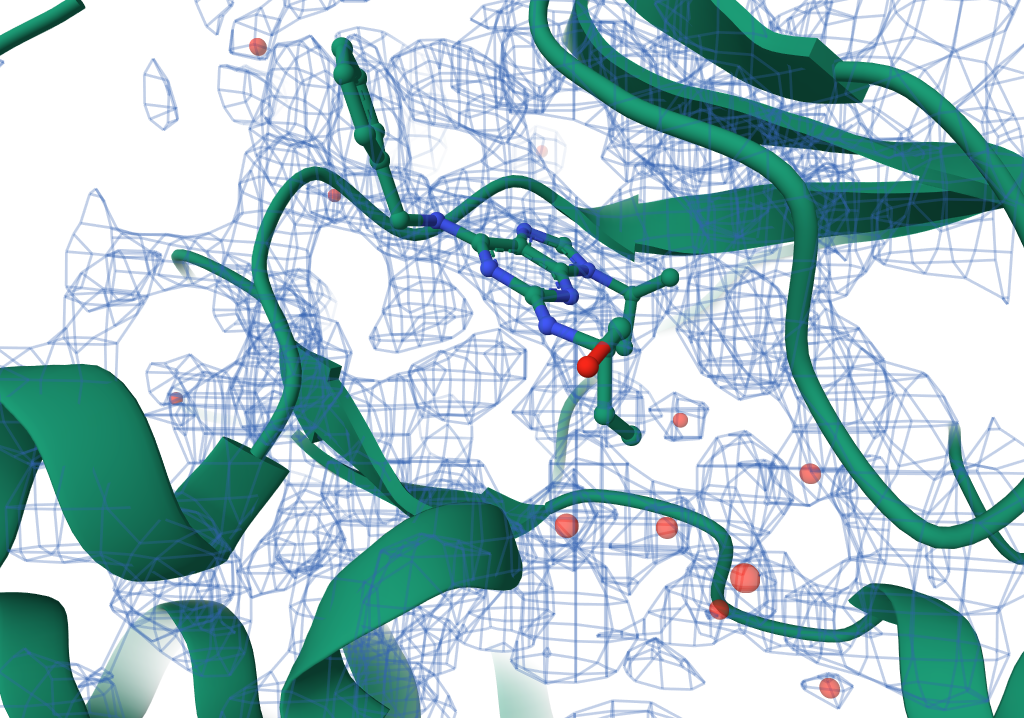
Aprendizado de Máquina Supervisionado II
Autor: Dr. Walter F. de Azevedo Jr.
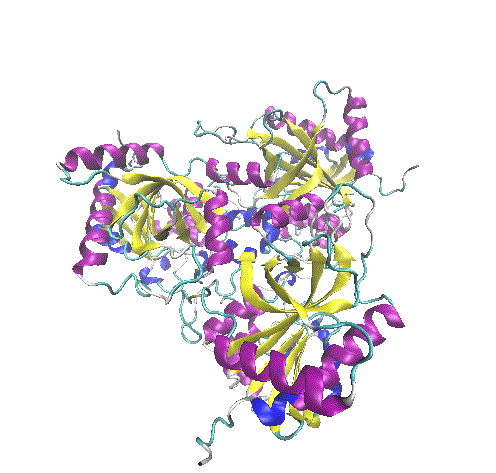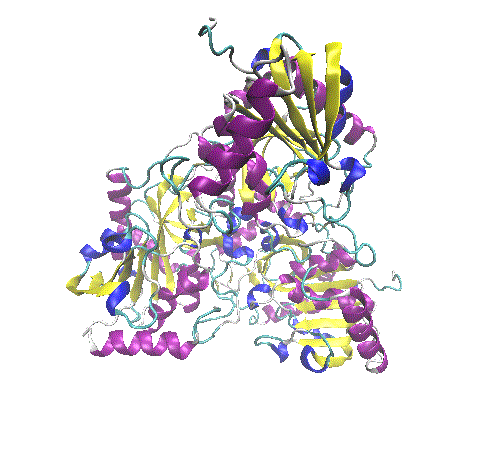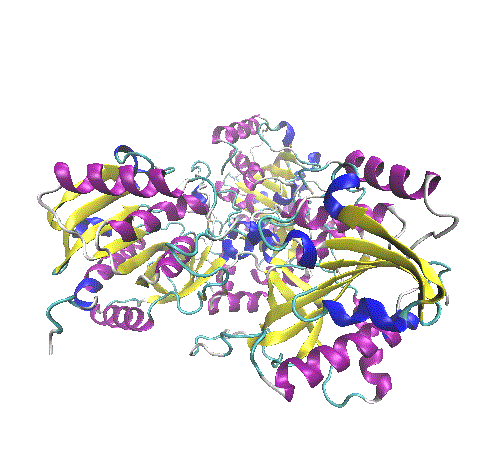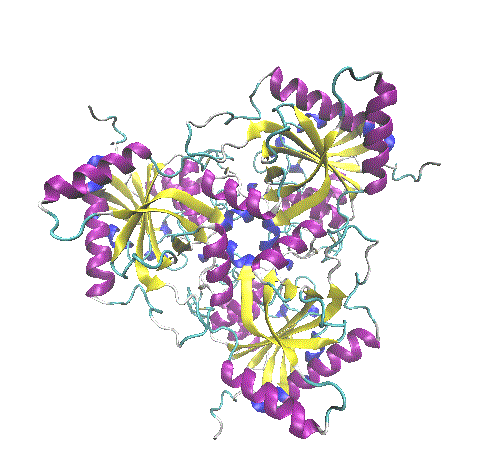
A quinase dependente de ciclina 2 (cyclin-dependent kinase 2, CDK2) está envolvida no ciclo celular. Foi identificada em aproximadamente 50 % dos tumores cancerígenos uma mutação no gene que codifica a proteína p53. Esta proteína é um fator de transcrição da proteína p21, uma proteína inibidora de CDK2. A inibição da CDK2 suspende a progressão do ciclo celular e pode levar à apoptose. No caso de células cancerígenas, a inibição da CDK2 evita a propagação do tumor. A partir desse conhecimento, começou-se a investigar a interação da CDK2 com inibidores não proteicos, como a molécula de roscovitina mostrada em complexo com a CDK2 na figura ao lado. Na aula de hoje, veremos simulações de docagem molecular contra essa proteína. Palavras-chave: Docking; molecular Docking; docagem; docagem molecular; computação bioinspirada; computação biologicamente inspirada; algoritmo evolucionário; algoritmo genético; algoritmo de evolução diferencial; string binária; problemas de otimização; cyclin-dependent kinase 2; CDK2; quinase dependente de ciclina 2; Molegro Virtual Docker; aprendizado de máquina; modelo de regressão linear; modelo de aprendizado de máquina; métricas; poder de previsão. |

|
|
Aprendizado de Máquina Supervisionado III
Autor: Dr. Walter F. de Azevedo Jr.
O uso da natureza como fonte de inspiração para o desenvolvimento de algoritmos tem gerado metodologias computacionais para solução de problemas de otimização. Quando usamos a biologia como inspiração para o desenvolvimento de algoritmos estamos na seara da computação biologicamente inspirada, ou simplesmente bioinspirada. Hoje veremos os algoritmos evolucionários que usam como inspiração as ideias da evolução de Darwin. Estudaremos os algoritmos genéticos e o algoritmo de evolução diferencial. Veremos com podemos usar o algoritmo de evolução diferencial para a simulação de docagem molecular. Aplicaremos a docagem molecular com evolução diferencial para a quinase dependente de ciclina 2. Em seguida iremos preparar dados para a construção de modelos e aprendizado de máquina para previsão da afinidade por CDK2. A partir desses dados mostraremos como construir um modelo para previsão da inibição da CDK2. Palavras-chave: Docking; molecular Docking; docagem; docagem molecular; computação bioinspirada; computação biologicamente inspirada; algoritmo evolucionário; algoritmo genético; algoritmo de evolução diferencial; string binária; problemas de otimização; cyclin-dependent kinase 2; CDK2; quinase dependente de ciclina 2; Molegro Virtual Docker; aprendizado de máquina; modelo de regressão linear; modelo de aprendizado de máquina; métricas; poder de previsão. |

|
|
Aprendizado de Máquina Supervisionado IV
Autor: Dr. Walter F. de Azevedo Jr.
Nesta aula mostraremos como métodos de aprendizado de máquina supervisionados podem contribuir para o desenvolvimento de pesquisas com foco na descoberta de fármacos. A partir das informações sobre a estrutura tridimensional de proteínas relacionadas a alguma patologia, podemos usar as informações experimentais para treinarmos modelos de aprendizado de máquinas. Uma vez treinados e validados, esses modelos são usados para busca por moléculas que podem apresentar ação farmacológica. A partir da analogia chave-fechadura, podemos pensar que os métodos de aprendizado de máquina nos auxiliam na busca de novas chaves (fármacos em potencial) que se encaixam numa dada fechadura (parte da proteína que interage com o fármaco). Iremos apresentar o conceito de espaço de funções escores e como usá-lo para construir modelos de aprendizado de máquina direcionados para proteínas de interesse. Palavras-chave: Docking; molecular Docking; docagem; docagem molecular; espaço químico; espaço de proteínas; espaço de funções escores; protein data bank; cristalografia; difração de raios X; mapa de densidade eletrônica; coordenadas atômicas; cyclin-dependent kinase 2; CDK2; quinase dependente de ciclina 2; Molegro Virtual Docker; aprendizado de máquina; modelo de regressão linear; modelo de aprendizado de máquina; métricas; poder de previsão. |
|



 SAnDReS: Statistical Analysis of Docking Results and Scoring functions
SAnDReS: Statistical Analysis of Docking Results and Scoring functions


 Taba: A Tool to Analyze the Binding Affinity
Taba: A Tool to Analyze the Binding Affinity